 |
||||
|
← |
Д.А. Шабанов, М.А.Кравченко. Статистический анализ данных в зоологии и экологии |
→ |
||
|
Тема 6. Сравнение распределений |
||||
|
Биостатистика-09 |
Биостатистика-11 |
|||
6.1. Примеры проблем, требующих сравнения распределений
Вероятно, сравнение распределений — столь же частая, как и сравнение значений, задача биологических исследований. В этом тексте тема сравнения распределений затрагивалась, как минимум, три раза.
Во-первых, примером сравнения распределений является сравнение выборок по критерию Фишера, описанное в пункте 4.3 (см. рис. 4.3.1). Это — параметрическое сравнение, так как распределения в этом случае сравниваются по одному параметру, основанному на предположении о том, что обе выборки имеют нормальное распределение.
Во-вторых, процедура сравнения распределений методом  Пирсона автоматически вызывается при сравнении выборок по Краскелу-Уоллису, описанной в пункте 4.11 (см. рис. 4.11.3). Это вполне логично: если мы хотим установить, к одной или нескольким генеральным совокупностям принадлежит несколько выборок, мы можем использовать их сравнение и по абсолютным значениям (сравнение средних или медиан), и по характеру их распределений.
Пирсона автоматически вызывается при сравнении выборок по Краскелу-Уоллису, описанной в пункте 4.11 (см. рис. 4.11.3). Это вполне логично: если мы хотим установить, к одной или нескольким генеральным совокупностям принадлежит несколько выборок, мы можем использовать их сравнение и по абсолютным значениям (сравнение средних или медиан), и по характеру их распределений.
В-третьих, мы уже рассмотрели ситуации, в которых приходится проверять распределения выборок на нормальность. Эта проверка имеет смысл во многих случаях, когда надо выбрать, параметрические или непараметрические методы следует использовать. Простой способ такой проверки описан в пункте 5.2 (см. рис. 5.2.1).
Как указывалось ранее (см. пункт 1.5), далеко не все признаки выражаются числом из непрерывного (метрические) или дискретного (меристические) числового ряда. Многие важные для биологического исследования признаки носят качественный (политомический или дихотомический), а также ранговый характер. Чтобы сравнить выборки по таким признакам, необходимо сравнение распределений. Приведем примеры нескольких задач, требующих использование методов, которые рассматриваются в этой теме:
— отличаются ли соседствующие локальные популяции по частотам характерных фенов (единичных качественных внешних признаков);
— отличаются ли представители разных полов по склонности к определенному заболеванию;
— действительно ли использование какого-то способа лечения приводит к более частому выздоровлению пациентов;
— зависимо или нет варьируют в изученной совокупности объектов два (или большее количество) качественных признаков.
Начнем рассмотрение методов сравнения распределений с использования критерия  Пирсона для решения последней из перечисленных задач.
Пирсона для решения последней из перечисленных задач.
6.2. Определение связи качественных признаков с помощью кросстабуляции
Обратимся к нашему файлу Pelophylax_example.sta. В нем рассмотрено 57 лягушек, относящихся к 5 разным генотипам и 2 разным полам. Распределение по полу и генотипу изученных лягушек не равномерное: одни сочетания встречаются чаще, другие — реже. Отражает ли наблюдаемое распределение связь между полом и генотипом? Можно ли предположить, что для одних генотипов у нас статистически значимо чаще попадались самки, а для других — самцы?
Чтобы ответить на этот вопрос, надо определиться с тем, какое распределение мы бы считали равномерным. То, в котором все 10 (2×5) возможных сочетаний пола и генотипа представлены с равной (или приблизительно равной частотой)? Но, весьма вероятно, некоторые генотипы (например, LLR) встречаются реже, а некоторые (как RR) — чаще. Как нам построить исследование, чтобы разная представленность генотипов и разная представленность полов в нашей выборке не мешала нам получить ответ на заданный вопрос?
Для ответа на этот вопрос откроем файл Pelophylax_example.sta и пройдем по пути Statistics / Basic Statistics and Tables / Tables and banners.
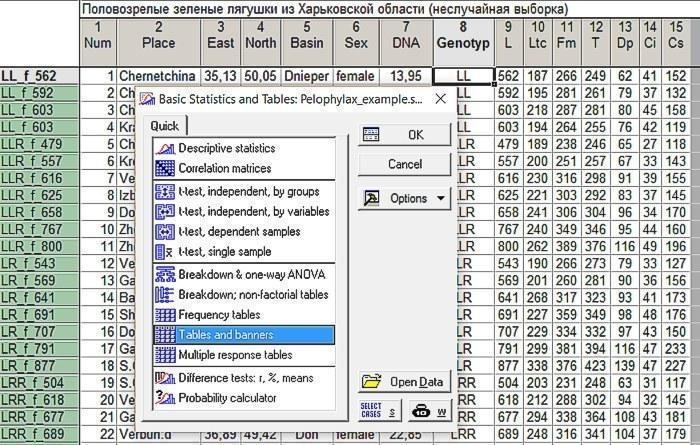
Рис. 6.2.1. Удобный диалог для сравнения распределений находится здесь
Мы попадем на вкладку Crosstabulation Tables. Выберем там переменные, соответствующие полу и генотипу.
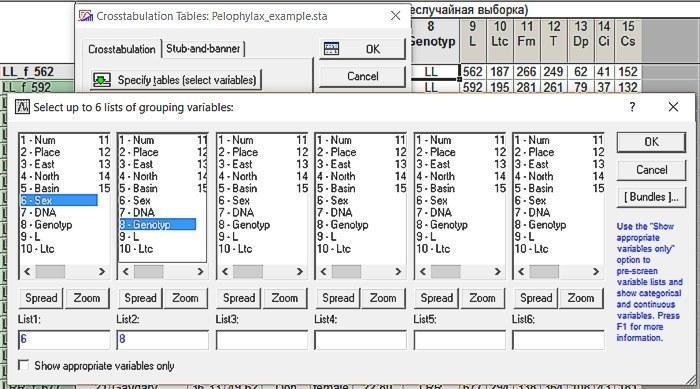
Рис. 6.2.2. Выбор переменных, связь между которыми надо проанализировать
В окне Crosstabulation Tables Results снимем галочку Highlight counts, которая позволяет выделить красным цветом значения в тех ячейках, куда попало относительно много наблюдений (по умолчанию — 10). Поставим галочки в окошке Expected frequencies, выводящем отдельное окно с ожидаемыми частотами (при наблюдаемых краевых суммах), а также в окошке Pearson & M-L Chi-square, которое вызывает сравнение наблюдаемых и ожидаемых частот по методу 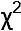 .
.
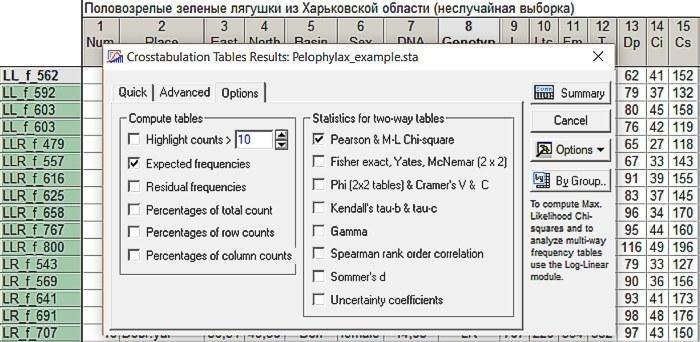
Рис. 6.2.3. Настройки, требуемые для сравнения ожидаемого и наблюдаемого распределений
Нажав на кнопку Summary, мы получим сразу две таблицы.
Рис. 6.2.4. Результат анализа. Выше — наблюдаемое распределение, ниже — ожидаемое (при тех же краевых суммах). Убедитесь, что краевые суммы (суммы элементов в каждой строке и каждом столбце) не изменились!
Интерпретация первой из полученных таблиц очень проста. Это наблюдаемые частоты разных сочетаний двух выбранных переменных. Той переменной, которая была выбрана первой, соответствуют строки этой таблицы, а второй переменной соответствуют столбцы. Кстати, с помощью обсуждаемого диалога можно изучать взаимосвязь также и трех или большего количества переменных, однако интерпретация таких многовходовых таблиц оказывается непростым делом (попробуйте и убедитесь сами!).
В таблице наблюдаемых частот посчитаны краевые суммы. В нашем случае это общее количество самок и самцов, а также особей различных генотипов. Обратите внимание: лягушек с генотипом LL исследовано 5 штук. Общее соотношение самок и самцов в нашей выборке 31:26. Если бы у особей с генотипом LL наблюдалось то же самое соотношение, мы могли бы ожидать у них соотношения приблизительно 2,7:2,8. С другой стороны, общее распределение генотипов в нашей выборке 5:11:14:13:14. Если бы особи были распределены равномерно, те же самые соотношения долей наблюдались бы и у самок, и у самцов.
Итак, таблица, содержащая Expectad Frequencies строится на основании эмпирически зарегистрированных краевых сумм, на основании предположения о том, что значения рассматриваемых переменных распределены все связи друг с другом. Распределения генотипов для разных полов соответствуют распределению генотипов во всей выборке; распределения полов для разных генотипов соответствуют распределению полов для всей выборки. Эта таблица дает ожидаемое распределение полов в том случае, если нулевая гипотеза (постулирующая отличие связи между переменными) верна.
Нам осталось сравнить наблюдаемое и ожидаемое распределение. В таблице это сделано с использованием критерия  (или критерия согласия), предложенного Карлом Пирсоном в 1900 году. Историки науки высказывали мысль, что статья, в которой Пирсон предложил этот критерий, открыла новый век в истории статистики. Не вдаваясь в детали, скажем, что Пирсон исследовал, насколько сильно распределение выборки может отличаться от распределения генеральной совокупности, из которой оно получено. Мера различия двух распределений вычисляется на основании суммы квадратов различий наблюдаемых и ожидаемых частот (в нашем случае — значений в ячейках двух таблиц, показанных на предыдущем рисунке). Естественно, "вес" этой меры различия распределений зависит от количества степеней свободы (количества ячеек, значения в которых можно изменять независимо). Эта мера различия распределений названа
(или критерия согласия), предложенного Карлом Пирсоном в 1900 году. Историки науки высказывали мысль, что статья, в которой Пирсон предложил этот критерий, открыла новый век в истории статистики. Не вдаваясь в детали, скажем, что Пирсон исследовал, насколько сильно распределение выборки может отличаться от распределения генеральной совокупности, из которой оно получено. Мера различия двух распределений вычисляется на основании суммы квадратов различий наблюдаемых и ожидаемых частот (в нашем случае — значений в ячейках двух таблиц, показанных на предыдущем рисунке). Естественно, "вес" этой меры различия распределений зависит от количества степеней свободы (количества ячеек, значения в которых можно изменять независимо). Эта мера различия распределений названа 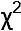 . Для разных степеней свободы построены распределения вероятностей
. Для разных степеней свободы построены распределения вероятностей 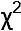 ; зная значение этой величины, зарегистрированное в нашем сравнении, мы можем определить вероятность его случайного возникновения (т.е. вероятность нулевой гипотезы).
; зная значение этой величины, зарегистрированное в нашем сравнении, мы можем определить вероятность его случайного возникновения (т.е. вероятность нулевой гипотезы).
Итак, в нашем сравнении вычисленное значение критерия 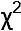 — 2,72, а количество степеней свободы df=4. Почему именно 4? Представьте себе, мы имеем таблицу с заданными краевыми частотами. В одну из ячеек, соответствующих самкам или самцам первого генотипа, мы можем вписать любое число, не превышающее количество представителей этого генотипа (и общее количество представителей этого пола во всей выборке); этим мы сразу определим и значение второй ячейки (оно высчитывается как общая численность представителей данного генотипа, из которой вычтено количество представителей первого пола данного генотипа). То же самое мы можем сделать со вторым, третьим и четвертым генотипами. После этого численность представителей обоих полов последнего, пятого генотипа уже окажется жестко заданной (количество самок вычисляется вычитанием из общего количества самок количества самок первого, второго, третьего и четвертого генотипов). Итак, в таблице 2×5 мы можем независимо менять 4 значения, т.е. df=4.
— 2,72, а количество степеней свободы df=4. Почему именно 4? Представьте себе, мы имеем таблицу с заданными краевыми частотами. В одну из ячеек, соответствующих самкам или самцам первого генотипа, мы можем вписать любое число, не превышающее количество представителей этого генотипа (и общее количество представителей этого пола во всей выборке); этим мы сразу определим и значение второй ячейки (оно высчитывается как общая численность представителей данного генотипа, из которой вычтено количество представителей первого пола данного генотипа). То же самое мы можем сделать со вторым, третьим и четвертым генотипами. После этого численность представителей обоих полов последнего, пятого генотипа уже окажется жестко заданной (количество самок вычисляется вычитанием из общего количества самок количества самок первого, второго, третьего и четвертого генотипов). Итак, в таблице 2×5 мы можем независимо менять 4 значения, т.е. df=4.
Как это отражено на предыдущем рисунке, связь между рассматриваемыми переменными в нашем примере оказалась незначимой (p=0,686; мы имеем тело с тем случаем, когда Statistica не ставит 0 перед десятичным разделителем). Итак, материал в нашем исследовании представлен неравномерно, но значимой связи между полом и генотипом мы не зарегистрировали.
6.3. Сравнение распределений с помощью модуля непараметрической статистики
В предыдущем пункте мы приняли зарегистрированные нами краевые частоты (общее количество самцов, самок, а также представителей разных генотипов) в нашей выборке как заданные. Мы предположили, что они отражают разное количество представителей разных полов и разных генотипов в той генеральной совокупности, из которой мы получили нашу выборку. Однако в некоторых случаях имеет смысл иная постановка вопроса. А можем ли мы на основании нашей выборки предположить, что разные полы и генотипы распределены в генеральной совокупности, которую мы изучаем, неравномерно? Для этого нам надо сравнить зарегистрированное распределение с иным, где в каждой ячейке находится одинаковое количество особей (легко понять, как его определить: 57/10=5,7). Для этого нам придется сделать файл с таким распределением.
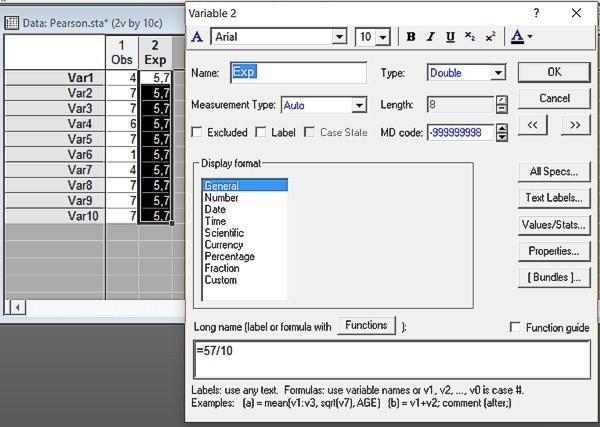
Рис. 6.3.1. В первом столбце приведены зарегистрированные частоты, во втором — ожидаемые частоты в том случае, если численность представителей обоих полов (а также разных генотипов) одинакова
Для того способа анализа, к которому мы планируем приступить, важно, чтобы общая численность обоих распределений была одинакова. В нашем случае это условие выполняется: мы сгенерировали ожидаемое распределение в пересчете на зарегистрированное количество особей. Тем же способом можно сравнивать и два эмпирических распределения, только при этом нужно пересчитать большее так, чтобы количество наблюдений в нем соответствовало меньшему.
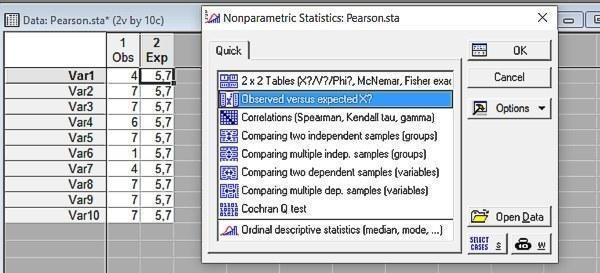
Рис. 6.3.2. Используемый нами метод относится к числу непараметрических
Осталось указать, какие распределения мы сравниваем, и получить результат.
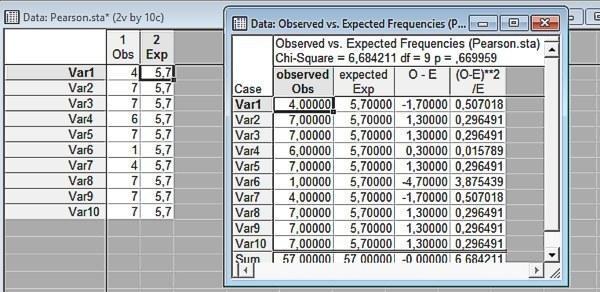
Рис. 6.3.3. Результат сравнения эмпирического и расчетного распределений. Разница незначима
На последнем рисунке мы можем увидеть, как происходит вычисление статистики 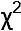 . Для каждой пары ячеек (для каждой категории сравниваемых нами распределений) определяется их разница, которая возводится в квадрат и делится на количество сравниваемых пар. Сумма этих величин и является величиной
. Для каждой пары ячеек (для каждой категории сравниваемых нами распределений) определяется их разница, которая возводится в квадрат и делится на количество сравниваемых пар. Сумма этих величин и является величиной 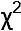 . Обратите внимание: величина
. Обратите внимание: величина 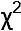 в данном случае выше, чем в предыдущем расчете, а значимость меньше. Почему? Потому что выше количество степеней свободы. В данном случае, зная общее количество наблюдений, мы можем независимо менять (в определенных пределах, естественно) значения в 9 ячейках.
в данном случае выше, чем в предыдущем расчете, а значимость меньше. Почему? Потому что выше количество степеней свободы. В данном случае, зная общее количество наблюдений, мы можем независимо менять (в определенных пределах, естественно) значения в 9 ячейках.
Итак, мы не нашли подтверждений предположению, что пола и генотипы лягушек распределены неравномерно (а в прошлом пункте — что они связаны друг с другом). На основании рассмотренных данных у нас нет достаточных причин отвергать нулевую гипотезу. Важное дополнение заключается в том, что для рассматриваемой проблемы известно, что нулевая гипотеза неверна. На территории изучения частоты разных генотипов неравны; для некоторых генотипов достаточно часто наблюдается преобладание самом или самцов. Однако эти утверждения делаются не на основании данных, находящихся в файле Pelophylax_example.sta, а с учетом иных, значительно более представительных исследований.
Таким образом, в нашем случае нулевая гипотеза не отвергнута. Как и во всех остальных случаях, это не означает, что она верна.